Parole chiave
Collezione di genotipi di frumento duro utilizzata nel progetto SURF
Le collezioni di germoplasma costituite dalle comunità scientifiche sono una risorsa primaria per esaminare la diversità genetica di specie vegetali e per identificare nuovi caratteri di interesse che possono essere utilizzati nel miglioramento genetico delle piante di interesse agrario, al fine di creare nuove varietà migliorate per la nutrizione umana, in un’ottica di sicurezza alimentare globale, e per un’agricoltura più sostenibile [1,2].
Il frumento duro [Triticum turgidum L. ssp. durum (Desf.) Husn.] è una delle colture più importanti per la nutrizione umana a livello mondiale, con una produzione annuale di più di 40 milioni di tonnellate [3]. Il frumento è l’ingrediente base per i cibi principali della tradizione mediterranea, come la pasta, il cous cous, il burghul. Il frumento duro si è evoluto dal farro [T. turgidum ssp. dicoccum (Schrank ex Schübl.) Thell.], che a sua volta si è originato dal farro selvatico [T. turgidum ssp. dicoccoides (Körn. ex Asch. & Graebn.) Thell.] nella Mezzaluna Fertile, circa 10000 anni fa [4,5].
Recentemente, il genoma del frumento duro è stato sequenziato, grazie allo sforzo congiunto di diversi gruppi di ricerca a livello internazionale, tra cui l’Italia [6]. Questo grande risultato scientifico ha dato nuovo impulso alla ricerca sul frumento duro, grazie alla disponibilità di dati genetici e genomici.
La collezione di frumento duro utilizzata nel progetto SURF è composta da più di 200 genotipi, tra cui 195 genotipi selvatici. Ottanta di questi provengono dalla collezione “single seed descent” (SSD) del CNR [7]. Questi genotipi sono stati selezionati da una collezione più ampia, la cosiddetta Global Durum Panel, formata da più di 1000 genotipi di frumento duro provenienti da tutto il mondo, che sono stati selezionati dalla comunità scientifica internazionale che studia la genetica del frumento duro [8].
Per il progetto SURF sono stati selezionati principalmente genotipi selvatici, in quanto questi genotipi possiedono un genoma che non è stato domesticato dall’uomo e potrebbero possedere caratteri di resistenza a malattie, tra cui quelle di origine virale, non ancora noti. In figura 1 è visibile l’origine geografica dei genotipi della collezione. Le zone maggiormente rappresentate sono Asia Occidentale (56 genotipi), Nord Africa (36 genotipi), Italia (34 genotipi), Asia Centrale (26), Corno d’Africa (23 genotipi).
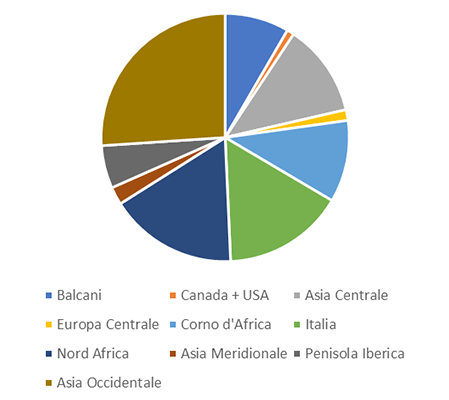
Figura 1: rappresentazione schematica della distribuzione geografica dei genotipi di frumento duro appartenenti alla collezione del progetto SURF.
I genotipi della collezione sono molto diversi tra loro per svariate caratteristiche evidenti, tra cui l’altezza, il colore delle foglie, il colore della spiga, la presenza e il colore delle reste, la tendenza all’allettamento, il tempo di fioritura e maturazione dei semi, come risulta evidente dalle fotografie qui riportate delle piante cresciute in campo. Questi caratteri mostrano chiaramente la variabilità genetica presente nella collezione selezionata.
Bibliografia:
1) Hufford MB, Berny Mier y Teran JC and Gepts P (2019). Crop Biodiversity: An Unfinished Magnum Opus of Nature. 70 727-751. 10.1146/annurev-arplant-042817-040240
2) Fowler C, and Hodgkin T (2004) PLANT GENETIC RESOURCES FOR FOOD AND AGRICULTURE: Assessing Global Availability. Annual Review of Environment and Resources 29:143-179. 10.1146/annurev.energy.29.062403.102203
3) Sall A, Chiari T, Legesse W, Seid-Ahmed K, Ortiz R, van Ginkel M, et al. (2019). Durum wheat (Triticum durum Desf.): origin, cultivation and potential expansion in Sub-Saharan Africa. Agronomy 9:263. 10.3390/agronomy9050263
4) Ozkan H, Brandolini A, Schafer-Pregl R, Salamini F (2002). AFLP analysis of a collection of tetraploid wheats indicates the origin of emmer and hard wheat domestication in southeast Turkey. Mol. Biol. Evol. 19 1797–1801. 10.1093/oxfordjournals.molbev.a004002
5) Dubcovsky J, Dvorak J (2007). Genome plasticity a key factor in the success of polyploid wheat under domestication. Science 316 1862–1866. 10.1126/science.1143986
6) Maccaferri M, Harris NS, Twardziok SO, Pasam RK, Gundlach H, Spannagl M, et al. (2019). Durum wheat genome highlights past domestication signatures and future improvement targets. Nat. Genet. 51 885–895. 10.1038/s41588-019-0381-3
7) Pignone D, De Paola D, Rapanà N, Janni M (2015) Single seed descent: a tool to exploit durum wheat (Triticum durum Desf.) genetic resources. Genet Resour Crop Evol 62: 1029–1035. doi:10.1007/s10722-014-0206-2.
8) Mazzucotelli E, Sciara G, Mastrangelo AM, Desiderio F, Xu SS, Faris J, et al. (2020) The Global Durum Wheat Panel (GDP): An International Platform to Identify and Exchange Beneficial Alleles. Front. Plant Sci. 11:569905. doi: 10.3389/fpls.2020.569905
GWAS (Genome Wide Association Study)
Uno studio di associazione è un’indagine, a livello di genoma, di varianti genetiche in diversi genotipi di una specie, allo scopo di verificare se alcune varianti sono associate ad uno specifico carattere di interesse [1,2]. Il primo dato che viene utilizzato per questo tipo di analisi sono precise informazioni genetiche, o marcatori molecolari. I marcatori molecolari più utilizzati per GWAS sono variazioni a livello di singola base nella sequenza del genoma, definite SNP (Single Nucleotide Polymorphism, cioè polimorfismo di singolo nucleotide).
La seconda fonte di informazione comprende la misurazione di un carattere fenotipico di interesse, che deve essere esplicitata sotto forma di numero.
Esempi di un carattere fenotipico per le piante sono l’altezza, il tempo di fioritura, la dimensione delle foglie o delle radici, la misurazione dei sintomi di un attacco da patogeno delle piante [3]. Una volta ottenute le informazioni genetiche e quelle fenotipiche di un alto numero di genotipi della stessa specie, si procede con associare il genotipo al fenotipo. L’analisi viene eseguita tramite software che utilizzano strumenti statistici di test delle ipotesi per dedurre l’associazione genotipo-fenotipo. Il risultato è la probabilità per ogni SNP di essere associato al fenotipo misurato. Questo viene anche rappresentato tramite il grafico Manhattan come mostrato in Figura 1.
Solitamente, vengono considerati gli SNP che hanno un valore superiore ad una certa soglia definita con metodi statistici (linea nera). Una volta identificati gli SNP che risultano più probabilmente associati al fenotipo di interesse, si procede a controllare i geni che si trovano nella regione del cromosoma vicina a questi SNP: vengono cioè cercati i possibili geni implicati nel cambiamento di fenotipo tra un genotipo e l’altro. Infine, si procede con la ricerca di variazioni genetiche precise di questi geni per capire le cause biologiche del fenotipo sotto esame.
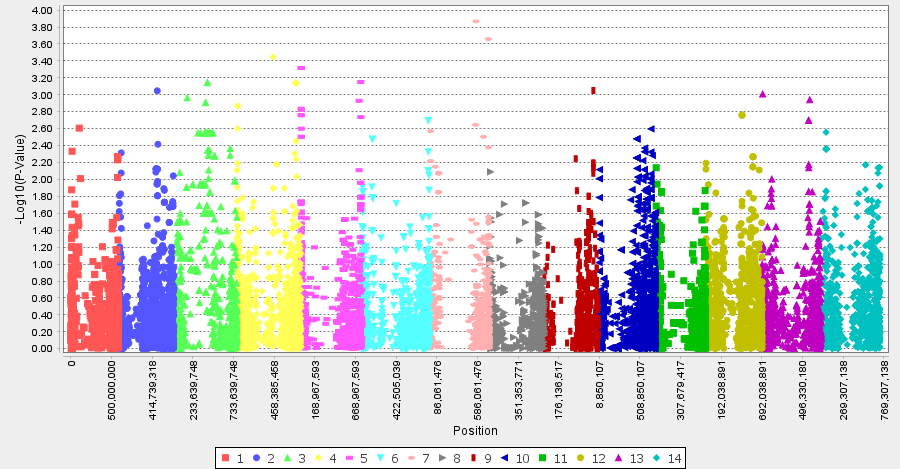
Figura 1. Grafico Manhattan di un’analisi di associazione. Sull’asse y la probabilità degli SNP sotto forma di valore negativo del logaritmo in base 10 (–log10(P-Value), e sull’asse x la posizione in paia di basi degli SNP sui singoli cromosomi della specie in esame. Ogni colore corrisponde ad un cromosoma, ogni puntino corrisponde a uno specifico SNP.
Bibliografia:
1) Klein RJ et al. (2005). Complement factor H polymorphism in age-related macular degeneration. Science 308 385–389. 10.1126/science.1109557
2) The Wellcome Trust Case Control Consortium (2007) Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls. Nature 447, 661–678.
3) Huang X and Han B (2014). Natural Variations and Genome-Wide Association Studies in Crop Plants. Annual Review of Plant Biology 65 531-551. doi.org/10.1146/annurev-arplant-050213-035715
Genome Editing
La tecnologia CRISPR (1), chiamata anche gene o genome editing, rappresenta una delle più recenti scoperte per il miglioramento genetico dei vegetali e si basa su un sistema molecolare di difesa che i batteri utilizzano per difendersi dai virus (2). L’importanza di questa tecnologia è evidenziata dal recente conferimento del premio Nobel alle due ricercatrici, la francese Emmanuelle Charpentier e la statunitense Jennifer A. Doudna, che per prime hanno contribuito allo sviluppo applicativo del CRISPR (3).
Ma in cosa consiste la tecnologia CRISPR?
IL CRISPR per funzionare necessita di due componenti principali: una proteina (CAS9), capace di tagliare il DNA, e una piccola molecola di RNA (l’RNA guida) che contiene una sequenza identica a una porzione del gene che intendiamo modificare: il nostro target. Quando questi due elementi vengono introdotti nella cellula, la sequenza di RNA guida la proteina CAS9 verso il DNA target permettendone la sua rottura in modo molto preciso. Il sistema di riparazione del DNA, presente naturalmente in tutte le cellule, pur tentando di riparare il danno creato dal CRISPR, agisce in modo impreciso introducendo delle mutazioni nel gene target. Il risultato finale consiste in cellule, che poi diventeranno piante, in cui un preciso carattere fenotipico, controllato dal gene che è stato mutato, risulterà inattivato (Figura 1).
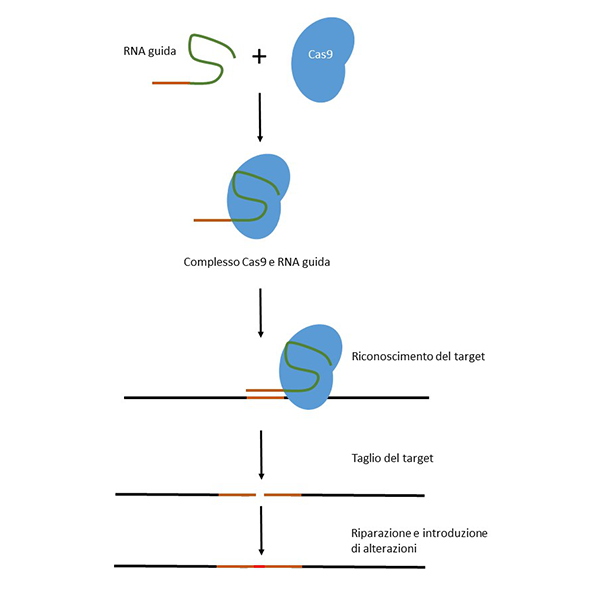
Figura 1. Rappresentazione schematica del meccanismo di gene editing. L’RNA guida e la proteina Cas9 interagiscono tra loro e questo complesso riconosce all’interno del genoma la sequenza target. La proteina Cas9 taglia il target e successivamente la sequenza di DNA tagliato viene riparato in modo non corretto. Queste imprecisioni determinano una variazione della sequenza del target e la sua inattivazione.
Attraverso la tecnologia CRISPR è possibile anche modificare in modo preciso un gene, al fine di migliorare un determinato carattere. Le mutazioni introdotte con il sistema CRISPR sono praticamente identiche a quelle che normalmente avvengono negli organismi durante la loro evoluzione, determinando l’attuale biodiversità esistente.
Il sistema CRISPR utilizzato per il miglioramento genetico delle piante, denominato New Breeding Techniques (NBT) o TEA (Tecnologie di Evoluzione Assistita), offre importanti vantaggi rispetto ai metodi usati precedentemente. Se paragonato alle metodologie di breeding classico, la tecnologia CRISPR risulta più veloce e più pulita in quanto la modifica del DNA è altamente specifica. Il sistema CRISPR rappresenta un avanzamento tecnologico rilevante anche rispetto agli organismi geneticamente modificati, in quanto non prevede necessariamente l’introduzione di geni estrani all’interno di un organismo: esistono infatti protocolli CRISPR che possono essere considerati OGM-free (4).
Inoltre, grazie alla conoscenza dei genomi di molte specie di interesse agrario, le potenzialità della tecnologia CRISPR sono davvero importanti in quanto qualsiasi gene presente in un organismo, di cui si conosca la funzione, può essere potenzialmente modificato per ottenere un carattere migliorativo per la produzione della specie di interesse. Questo permette di rispondere efficacemente e in tempi rapidi alla continua richiesta di piante sempre più performanti e adatte alle nuove realtà produttive fortemente influenzate dai recenti cambiamenti climatici.
In quest’ottica, all’interno del progetto SURF verrà impiegata la tecnologia CRISPR/Cas per modificare alcuni geni di frumento che favoriscono l’insorgenza di virosi, le quali determinano importanti perdite di produttività di questa coltura (5). Nel caso specifico del nostro progetto, questi geni che favoriscono l’infezione virale, una volta modificati, risulteranno non attivi inibendo l’attacco del virus.
1) FENG, Zhengyan, et al. Efficient genome editing in plants using a CRISPR/Cas system. Cell research, 2013, 23.10: 1229-1232.
2) BARRANGOU, Rodolphe, et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315.5819: 1709-1712.
3) JINEK, Martin, et al. A programmable dual-RNA–guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337.6096: 816-821.
4) ZHANG, Yi, et al. Efficient and transgene-free genome editing in wheat through transient expression of CRISPR/Cas9 DNA or RNA. Nature communications, 2016, 7.1: 1-8.
5) YANG, Ping, et al. PROTEIN DISULFIDE ISOMERASE LIKE 5-1 is a susceptibility factor to plant viruses. Proceedings of the National Academy of Sciences, 2014, 111.6: 2104-2109.
La Sensoristica in vivo per il monitoraggio precoce delle malattie
Uno dei punti chiave nello sviluppo di nuove varietà di frumento resistenti alle malattie e nello specifico alle virosi è la selezione di materiale genetico con incrementate capacità di difesa. La possibilità di identificare in stadi precoci genotipi resistenti alle virosi è alla base della selezione varietale. L’ausilio di biosensori può facilitare il compito del breeder e velocizzare il processo di selezione dei fenotipi resistenti.
Il CNR ha sviluppato un sensore in vivo, denominato Bioristor, in grado di rilevare i cambiamenti che avvengono nella linfa della pianta in seguito a stress di natura ambientale e in seguito ad attacco di patogeni [1,2]. Il Bioristor è un sensore organico elettrochimico (OECT) che viene inserito nel fusto della pianta. E’ composto da due fili tessili funzionalizzati con un polimero organico (PEDOT::PSS) chiamati gate e canale del sensore (Figura 1).
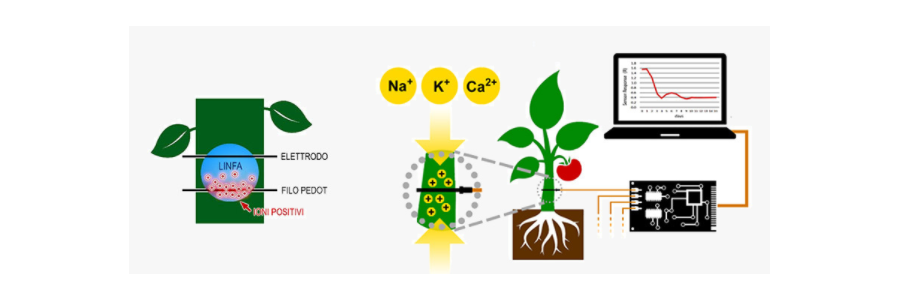
Figura 1: Principio di funzionamento del Bioristor.
In presenza di un potenziale applicato al gate, gli ioni positivi contenuti nella linfa vengono spinti nel canale che assorbendoli cambia la sua conducibilità. L’indice rilevato è la Risposta del sensore (R). IL Bioristor si è dimostrato molto efficace nel rilevare stadi precoci di stress idrico e di stress salino, anche in frumento (Figura 2) [3,4].

Figura 2: Il Bioristor in frumento.
1) Coppedè N., Janni M., Bettelli M., Maida C.L., Gentile F., Villani M., Ruotolo R., Iannotta S., Marmiroli N., Marmiroli M., et al. (2017). An in vivo biosensing, biomimetic electrochemical transistor with applications in plant science and precision farming. Sci. Rep. 7, 16195.
2) Vurro F., Janni M. (2020). Dar voce alle piante: il bioristor e il futuro dell’agricoltura. Sapere Sci. 6, 28–31.
3) Janni M., Coppede N., Bettelli M., Briglia N., Petrozza A., Summerer S., Vurro F., Danzi D., Cellini F., Marmiroli N. et al. (2019). In Vivo Phenotyping for the Early Detection of Drought Stress in Tomato. Plant Phenomics 2019, 1–10.
4) Vurro F., Janni M., Coppedè N., Gentile F., Manfredi R., Bettelli M., and Zappettini A. (2019). Development of an In Vivo Sensor to Monitor the Effects of Vapour Pressure Deficit (VPD) Changes to Improve Water Productivity in Agriculture. Sensors 19, 4667.
Resistenza ai virus
Le piante, come ogni altro organismo vivente, sono soggette a malattie causate da virus, per le quali non esistono prodotti curativi. Una situazione che quindi è assai diversa dalle malattie fungine, per le quali numerosi prodotti sono ampiamente utilizzati: basti pensare alla peronospora della vite. In assenza di fitofarmaci ad azione antivirale, la migliore arma di difesa contro i virus è la prevenzione attraverso l’uso di varietà resistenti.
Anche il grano duro, che rappresenta la principale coltura cerealicola italiana, con una superficie di circa 1,23 milioni di ettari nel 2019/2020 (dati Istat), è colpito da diverse malattie virali, che ne limitano la resa. Tra i virus che attaccano il frumento, il soil-borne cereal mosaic virus (SBCMV) è quello maggiormente presente nei nostri ambienti. Il SBCMV, appartenente al genere Furovirus [1], è caratterizzato da particelle rigide a forma bastoncellare, con una lunghezza di circa 160 e 300 nm (Figura 1). Uno dei primi paesi europei dove questo virus è stato rilevato è stata proprio l’Italia [2].
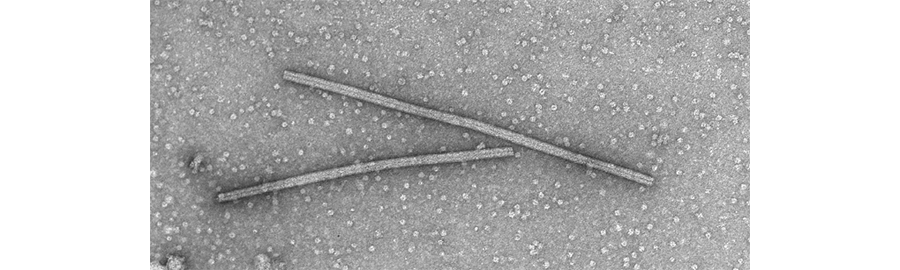
Figura 1: particelle virali di SBCMV visualizzate al microscopio elettronico.
Il SBCMV è trasmesso alle piante ospiti da un plasmodiophoromycota che vive nel terreno, Polymyxa graminis [3]. Questo protozoo è distribuito in tutto il mondo e si moltiplica principalmente nelle radici di specie erbacee e cerealicole. È un parassita intercellulare obbligato con spore di resistenza, nella quali il virus può rimanere vitale per oltre 15 anni, e zoospore primarie e secondarie mobili. Il vettore fungino fornisce protezione per il virus contro condizioni ambientali sfavorevoli e durante le rotazioni di colture non ospiti, inoltre consente al virus di persistere per diversi anni una volta che il campo è stato infestato [4,5,6] Data la non uniforme distribuzione del vettore in campo l’infezione si presenta a macchie più o meno estese che aumentano di dimensione col passare degli anni.
I sintomi del SBCMV sono visibili a fine inverno/ inizio primavera. Le piante si presentano con dimensioni ridotte e un mosaico fogliare con macchie clorotiche, prevalentemente di forma allungata, disposte parallelamente alle nervature (Figura 2).

Figura 2: piante di frumento duro (cv Claudio), inoculate con SBCMV. Dopo 30-35 giorni dall’inoculazione sono visibili i tipici sintomi del mosaico sulle foglie.
La risposta varietale varia da molto suscettibile a parzialmente resistente, ma una vera resistenza genetica non è ancora disponibile.
Ma che cos’è la resistenza di una pianta a un virus? La resistenza è la capacità della pianta di bloccare o ostacolare l’infezione. Il grado di resistenza varia in base alla combinazione ospite-patogeno. Il livello più elevato di resistenza è l’immunità, che si ha quando la pianta impedisce l’insediamento del patogeno. Una forma più blanda di resistenza è la tolleranza, in cui il patogeno invade i tessuti della pianta ma la pianta non risente molto del danno e in taluni casi non mostra sintomi.
Come si ottengono delle piante resistenti? I due approcci principali sono quello della genetica classica, che può essere accelerata grazie a metodiche di biologia molecolare, e quello biotecnologico, in cui si fa ricorso a specifiche tecniche di ingegneria genetica.
Il progetto SURF prevede, da un lato, di valutare il grado di resistenza di diverse varietà di frumento duro, attraverso l’utilizzo di una collezione di frumento duro costituita da più di 200 genotipi, tra cui 195 genotipi selvatici, e di identificare specifici geni che conferiscono resistenza, attraverso uno studio di associazione (o GWAS, Genome Wide Association Study, vedi sezione dedicata). Questi geni, presenti solo in alcune varietà di frumento resistenti, potranno poi essere introgrediti in varietà élite attraverso programmi di miglioramento genetico. Inoltre, il progetto SURF prevede lo sviluppo di linee resistenti in cui un gene di sensibilità risulti inattivato attraverso un approccio di genome editing, come descritto nella sezione dedicata.
1) Koenig, R., Huth, W., 2000. Nucleotide sequence analyses indicate that a furo-like virus from cereal, formerly considered to be a strain of Soil-borne wheat mosaic virus, should be regarded as a new virus species: Soil-borne cereal mosaic virus. J. Plant Dis. Protect. 107, 445–446.
2) Canova, A., 1964. Ricerche sulle malattie da virus delle Graminacee. I. Mosaico del Frumento transmissibile attraverso il terreno. Phytopathol. Medit. 3, 86–94.
3) Canova, A., 1966. Ricerche sulle malattie da virus delle Graminacee III. Polymyxa graminis Led. Vettore del virus del mosaico del Frumento. Phytopathol. Medit. 5, 53–58.
4) Braselton, J.P., 1995. Current status of plasmodiophorids. Crit. Rev. Microbiol. 21, 263–275
5) Kanyuka, K., Ward, E., Adams, M.J., 2003. Polymyxa graminis and the cereal viruses it transmits: a research challenge. Mol. Plant Pathol. 4, 393–406.
6) Linford, M.B., McKinney, H.H., 1954. Occurrence of Polymyxa graminis in roots of small grains in the United States. Plant Dis. Report. 38, 711–713.
I modelli predittivi
La possibilità di predire un evento futuro od un comportamento atteso sulla base di informazioni raccolte in momenti precedenti all’evento stesso rappresenta, insieme all’inferenza, uno dei due principali obiettivi della statistica. Negli ultimi anni l’utilizzo di modelli predittivi è diventato sempre più comune ed all’ordine del giorno in particolare grazie da un lato alla disponibilità di nuovi dati di diversa origine (e.g. dati produttivi raccolti in real-time, dati provenienti da analisi genomiche) e dall’altro grazie al concomitante sviluppo di processori sempre più potenti che permettono l’utilizzo di modelli anche molto complessi (Zeng and Lumley, 2018). A questo riguardo, anche il termine algoritmo è oggi entrato nel linguaggio comune, indicando con questo la sequenza di istruzioni eseguite per effettuare un determinato calcolo.
La figura 1 riassume in modo molto sintetico la struttura di un modello statistico di tipo predittivo. A sinistra abbiamo i nostri “input” che possono quindi essere dati eterogenei raccolti in campo od in laboratorio. A destra abbiamo il nostro “output” o predizione (malato si/no, livello produttivo, etc etc). In mezzo ci sono le istruzioni (algoritmo) che ci permettono di sviluppare il modello migliore e di predire con maggiore accuratezza il nostro output a partire dal nostro input.
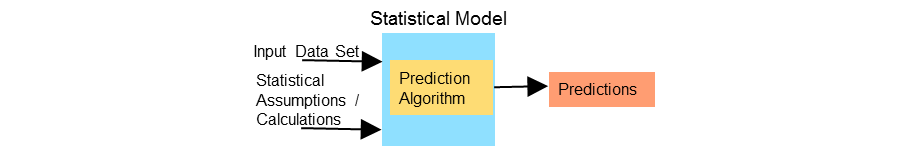
Figura 1 Struttura Base di un modello di predizione
Una delle difficoltà insite nello sviluppo di un modello predittivo risiede anche e soprattutto nella scelta dell’algoritmo giusto, scelta che dipende a sua volta sia dalla struttura dei dati disponibili ma anche dal tipo di risposta osservata. In questo senso lo sviluppo di un modello predittivo è un processo iterativo che cerca di minimizzare le differenze tra la nostra stima ed il dato reale.
L’applicazione di modelli predittivi nel mondo vegetale è anch’esso all’ordine del giorno spaziando dall’interazione pianta-patogeno (Sperschneider 2020) alla predizione di caratteri complessi (Montesinos-López et al, 2019) fino allo sviluppo di app per cellulari per l’identificazione di specie vegetali attraverso l’uso di immagini (https://identify.plantnet.org/, GBIF 2021).
1) Zeng ISL, Lumley T. Review of Statistical Learning Methods in Integrated Omics Studies (An Integrated Information Science). Bioinform Biol Insights. 2018 Feb 20;12:1177932218759292. doi: 10.1177/1177932218759292. PMID: 29497285; PMCID: PMC5824897.
2) Sperschneider J. Machine learning in plant-pathogen interactions: empowering biological predictions from field scale to genome scale. New Phytol. 2020 Oct;228(1):35-41. doi: 10.1111/nph.15771. Epub 2019 Mar 26. PMID: 30834534.
3) Montesinos-López OA, Martín-Vallejo J, Crossa J, Gianola D, Hernández-Suárez CM, Montesinos-López A, Juliana P, Singh R. A Benchmarking Between Deep Learning, Support Vector Machine and Bayesian Threshold Best Linear Unbiased Prediction for Predicting Ordinal Traits in Plant Breeding. G3 (Bethesda). 2019 Feb 7;9(2):601-618. doi: 10.1534/g3.118.200998. PMID: 30593512; PMCID: PMC6385991.
4) GBIF: The Global Biodiversity Information Facility (2021) What is GBIF?. Available from https://www.gbif.org/what-is-gbif
















